LSCGAT: Инструмент настраиваемого глобального выравнивания длинных последовательностей.
Справка
Возможности приложения можно продемонстрировать на следующем примере.
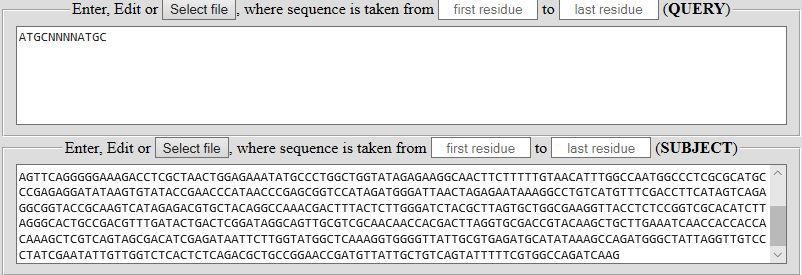
Найти вхождение или наиболее близкий аналог короткого биотехнологического паттерна ATGCNNNNATGC (QUERY) в длинной нуклеотидной последовательности (SUBJECT).
Для решения задачи надо учитывать следующие настройки в программе:
| Последовательности могут быть набраны, скопированы или введены из файла формата FASTA. Случайные последовательности могут быть сгенерированы и/или изменены с помощью отдельных сервисов. | |
 |
|
| Меню программы содержит выключатель панели настроек. По умолчанию программа согласована с программой BLAST Global Alignment. |  |
| Результаты включают в себя статистику совпадений, вес выравнивания, замер производительности (времена: общее, прямого и обратного хода), само выравнивание с координатами. При выравнивании двух случайных последовательностей при настройках по умолчанию реализуется близкое к 50% совпадение. | |
 |
|
| Выбор предустановленных настроек программы осуществляется с помощью выпадающего меню. |  |
| В матрице весов можно установить положительные значения за замену нуклеотида N на любой другой (последняя строка и последний столбец матрицы) – что позволяет не разрывать перетяжку NNNN. Другой способ запрета разрыва последовательности указан ниже в таблице. |  |
| При выравнивании короткой последовательности на длинную необходимо установить штрафы за концевые вставки равными 0 – что позволяет избавиться от влияния концов на результат. | 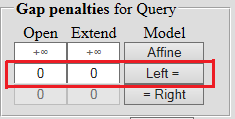 |
| Для запрещения разрыва в последовательности или вставки более одного символа можно установить бесконечный вес, оставив пустым соответствующее поле, в котором появится знак бесконечности. | 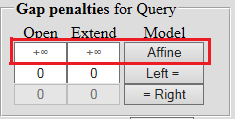 |
| Системы приоритетов обратного хода можно менять или установить случайную – что позволяет находить все варианты вхождения паттерна. | 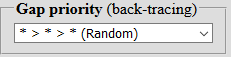 |
| Если на компьютере много ядер, то можно установить желаемое количество процессоров (по умолчанию - 4). Разбиение последовательностей на подпоследовательности осуществляется автоматически, но может быть задано и вручную. |  |
 SBARS.IMPB.RU
SBARS.IMPB.RU